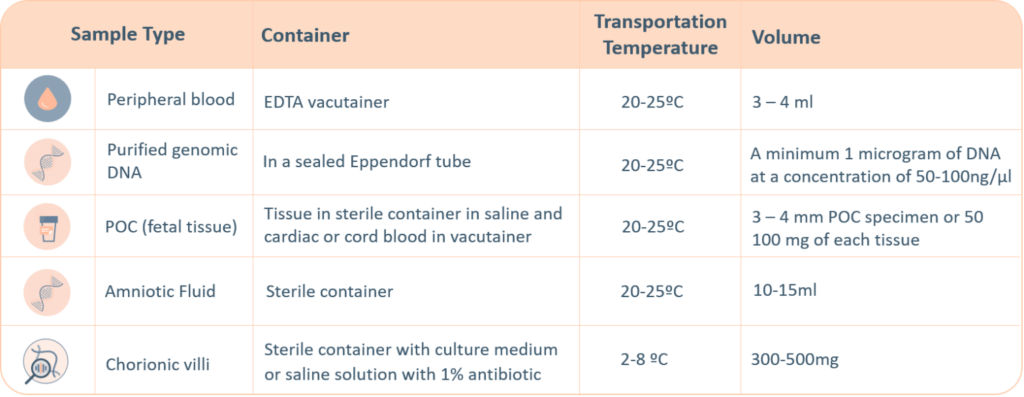
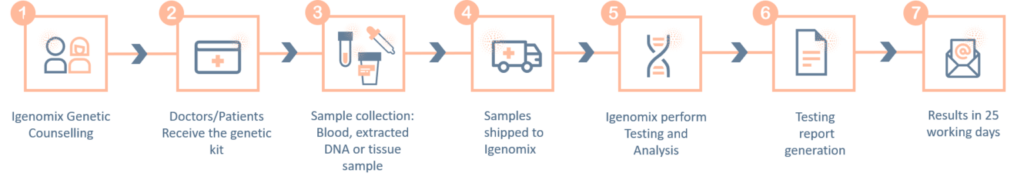
- El Microarray cromosómico (CMA), también conocido como array CGH (Hibridación Genómica Comparada), es un test diagnóstico que permite detectar, tanto grandes alteraciones cromosómicas numéricas (aneuploidías) como otras alteraciones submicroscópicas del número de copias (microdeleciones o microduplicaciones) a lo largo del genoma.
- CMA es la técnica gold standard para el análisis de deleciones y duplicaciones a lo largo de todo el genoma.
- CMA proporciona una resolución submicroscópica, permitiendo visualizar pequeñas regiones cromosómicas que el cariotipo no puede detectar.
- La sensibilidad del CMA depende del número de sondas moleculares utilizadas y de su cobertura a lo largo del genoma. Cuanto mayor sea el número de sondas (180K, 400K, 750K…), mayor será su sensibilidad.
CMA – Microarray cromosómico
CMA es la técnica de referencia para el análisis de CNVs (deleciones o duplicaciones) a nivel cromosómico